|
|
[论文笔记] U-Net: Convolutional Networks for Biomedical Image Segmentation
欢迎大家关注我的专栏,顺便点个赞~~~
个人心得:
- U-Net发表于2015年,用于医学细胞分割
- 编码器-解码器架构,四次下采样(maxpooling),四次上采样(转置卷积),形成了U型结构
- SGD+Momentum,损失函数为交叉熵
- 数据预处理使用了镜像边缘,可以更好细化边界信息
- 数据增加中有一个弹性形变,符合细胞本身的特性
- 可以应对小样本的数据集进行较快、有效地分割,能够泛化到很多应用场景中去
阅读背景
刚入门CV的小萌新,最开始看的都是目标检测的综述,但是由于才疏学浅,看不太懂,遂浅尝辄止。帮女票一直进行heasoft数据处理之余,意外看到一个遥感卫星图片中道路提取的课题,有点兴趣,遂开始研究。
因为意外参考了一个EPFL学生的代码,莫名其妙地走上了语义分割的道路,莫名其妙地使用了U-Net进行了分割,没错,全是意外。但在途中遇到了很多坑,看了很多讲解呀、样例代码呀,在细节上有很多问题,于是又仔细地开始阅读论文。
截图全部来自U-Net原文,http://arxiv.org/abs/1505.04597
本文作于2019年12月19日。
摘要
There is large consent that successful training of deep networks requires many thousand annotated training samples.
提到了卷积神经网络(CNN)要求数以千计的有标签数据,这已经成为了共识。
In this paper, we present a network and training strategy that relies on the strong use of data augmentation to use the available annotated samples more efficiently.
本文提出了一种网络结构和一种高效利用有标签数据的策略。
The architecture consists of a contracting path to capture context and a symmetric expanding path that enables precise localization. We show that such a network can be trained end-to-end from very few images and outperforms the prior best method (a sliding-window convolutional network) on the ISBI challenge for segmentation of neuronal structures in electron microscopic stacks.
这种U型结构,下采样的过程可以捕捉语义信息,与之对应的上采样过程可以进行精确地定位。这个网络可以利用非常少的图片进行端到端训练,但是效果缺超过了此前的最好的模型——基于滑动窗口的CNN(这个具体是啥,留坑)。
Using the same net- work trained on transmitted light microscopy images (phase contrast and DIC) we won the ISBI cell tracking challenge 2015 in these categories by a large margin. Moreover, the network is fast. Segmentation of a 512x512 image takes less than a second on a recent GPU. The full implementation (based on Caffe) and the trained networks are available at http://lmb.informatik.uni-freiburg.de/people/ronneber/u-net.
该模型参与多个竞赛,效果领先一大截,速度快,有Caffe的开源实现版本。(pytorch版本直接搜GitHub即可)
引言
总起CNN的发展历史和之前为什么没有发展起来的原因,提到CNN在图像分类中的应用。
However, in many visual tasks, especially in biomedical image processing, the desired output should include localization, i.e., a class label is supposed to be assigned to each pixel.
基本上说明了语义分割的意思——对每一个像素进行分类。
- 网络结构
作者总结了上文提到的滑动窗口的不足,然后扔出了自己的模型,一种“elegant"的全卷积网络。
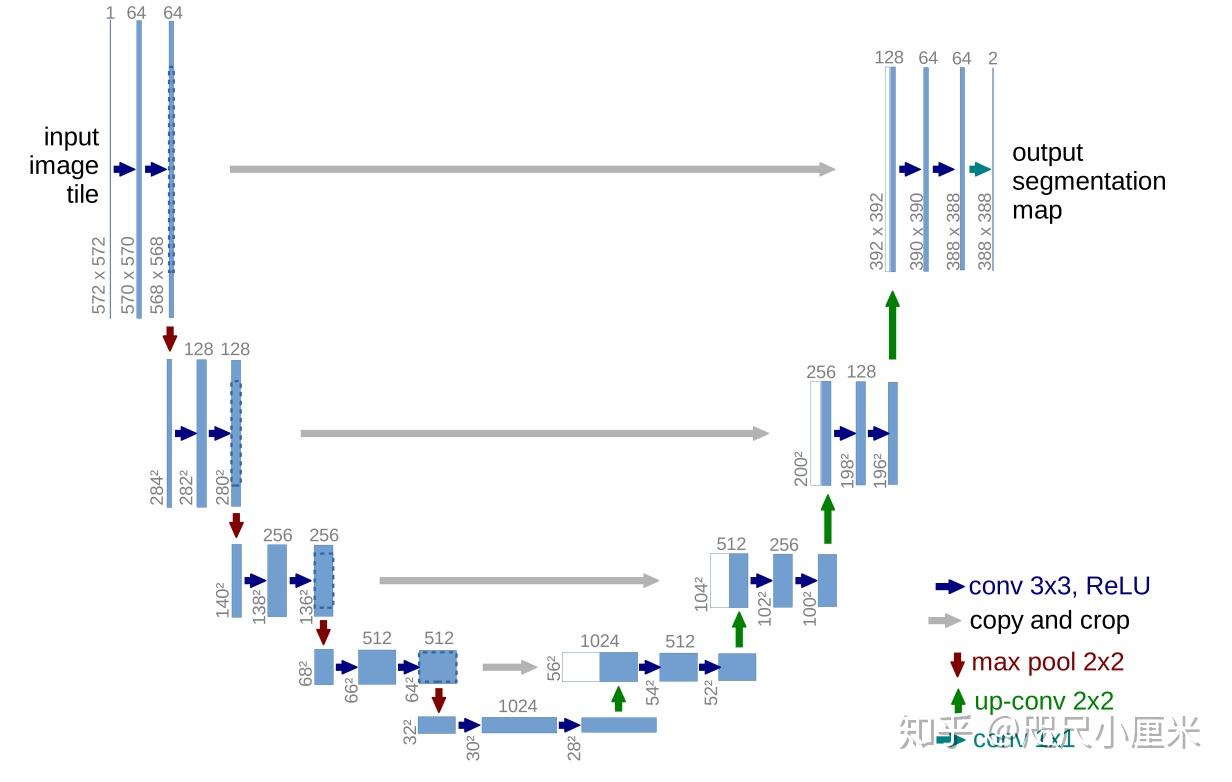
One important modification in our architecture is that in the upsampling part we have also a large number of feature channels, which allow the network to propagate context information to higher resolution layers. As a consequence, the expansive path is more or less symmetric to the contracting path, and yields a u-shaped architecture.
个人的理解是保持较多的卷积核数量,可以更好地捕捉和传播语义信息。
- 镜像边缘
This strategy allows the seamless segmentation of arbitrarily large images by an overlap-tile strategy.
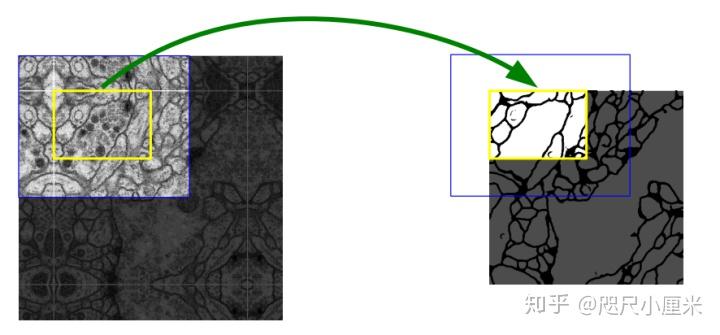
这是我之前一直没搞明白的地方,就是为啥要这样?平常训练输入输出的feature map都是尺寸大小一致,但是为什么分割黄框要输入蓝框的数据(更大尺寸的数据)呢? 因为论文中的U-Net的输入和输出尺寸是不一致的,上图中输入时572*572,但是输出是392*392,也正符合训练黄框要蓝框大小的输入,那么问题又来了,为什么要这样做呢? 我的理解是,输入的图像其实很大,由于显存的限制,我们不能一次性训练一张很大的图,所以必然是要切割图像的。但是切割图像,分割之后合并,边缘情况可能存在误差,比如连接不上等情况。但是,如果利用周围多一圈的语义信息,一定程度上能够很好地处理这种边缘的问题,因此这样做。 那么,新的问题诞生了,角落区域的图像,可能没有周围一圈,怎么办?作者给出的做法就行,做镜像。仔细观察图,我们就会发现,图中上面和左边,有部分是关于黄框对称的,大概就是这个意思。
As for our tasks there is very little training data available, we use excessive data augmentation by applying elastic deformations to the available training images. This allows the network to learn invariance to such deformations, without the need to see these transformations in the annotated image corpus. This is particularly important in biomedical segmentation, since deformation used to be the most common variation in tissue and realistic deformations can be simulated efficiently. 大概意思就是通过弹性形变增强数据,能加强模型的鲁棒性,能够学习到ceil中一些不变的特征,值得一提的是,弹性形变也是细胞本身具有的生理学特征。
网络结构

将输入进行2次3*3的卷积操作之后,进行一次2*2的最大池化,同时卷积核数目翻倍(1-64-128-256-512-1024),进行四次这样的down操作,然后2次3*3卷积,取一半的卷积核进行上采样(转置卷积),再剪切对应down层的feature map,和上采样得到的feature map拼接在一起,然后重复这样的过程。最后输出是深度为n_classes个feature map,进行分类。
这里最值得一提就是这个feature map拼接的操作,我认为,在在上采样的过程中会丢失部分语义特征,通过拼接的方式,可以恢复部分的语义信息,从而保证分割的精度。相似的,在FCN中(语义分割开山之作,我还没看),是通过feature map相加的方式来恢复语义信息的。
训练
常规训练,给出几个关键词——GPU,Caffe,momentum=0.99,cross entropy。 注意,这里的交叉熵是对像素级别进行分类的,值得一提的是,我用pytorch进行复现的时候,弄不清Crossentropyloss和BCEwithlogitsloss,总结的结果是,如果是二分类,用BCE就行了,输入(N,C,H,W),输出(N,1,H,W)的矩阵就行(值非1即0),若是多分类,输入(N,C,H,W),输出(N,n_classes,H,W),相当于对于每一个像素点进行一个one-hot表示。 这里有一个weight map,我搞不清作用,留坑,

Weight Map
实验
很之前的模型作对比,Warping Error,Rand Error,Pixel Error这三个指标不是很熟悉,留坑。
总结
主要提及了符合细胞生理特征的弹性形变——这种数据增强方式的意义,还有U-Net这个网络可以应对小样本的数据集进行较快、有效地分割,能够泛化到很多应用场景中去。 |
|